光作为一种清洁的、非侵害性的外源刺激手段,被广泛应用于在时间和空间维度上调节生物过程,高精度地实现对生命活动和过程的研究。实现生物过程的光调控主要有两种途径:光遗传学技术, 将一些吸收光后发生活性变化的光敏蛋白通过基因重组转入细胞,在神经生物学和细胞生物学研究中得到广泛应用;另一种途径是为体内具有生物功能的小分子、蛋白和核酸等活性物质装上光化学开关, 当这些活性物质被修饰上光敏感基团后,其活性可以被暂时封闭; 而在随后需要的时间点和空间位置光照切除光敏感基团或使其构型改变,恢复活性物质的生物功能[1-3]。本文主要针对第二种途径的光调控策略, 阐述近年来核酸光化学生物学的进展。
过去十几年中,随着测序技术的发展和新的寡聚核苷酸工具的开发,及核酸新功能的不断发现,对核酸功能的可控调节成为一个越来越吸引人的领域。在核酸中引入光敏感基团能够方便地通过光照时间、强度、波长等变量的改变实现对核酸功能的精确调控,因而受到研究者们的普遍重视。自1978年Kaplan等[4]首先合成了光敏感腺嘌呤三磷酸(ATP)后,研究者们陆续将光控策略应用到在生物体内发挥重要作用的各类核苷酸、寡聚核苷酸和DNA/RNA中。特别是在过去几年中,光控核酸及其在化学生物学中的应用取得了较大的发展,包括光敏核苷酸的应用、寡聚核苷酸光控策略的优化及其对基因表达和蛋白质功能的调控,以及DNA/RNA的光敏修饰策略等。
1 光敏感基团在化学生物学研究中应用较多的光敏感基团主要包括光致断裂基团和光致异构化基团[6](图 1)。光致断裂基团在光照下吸收能量后,会发生电子转移和能级跃迁,导致键的断裂,释放出被修饰的功能分子。邻硝基苄基(NPP)及其衍生物,如4, 5-二甲氧基-2硝基苄基(DMNB)和6-硝基-3, 4-亚甲二氧苄氧甲基(NPOM)等,因其合成和使用方便、稳定性好,目前仍是应用最广的一类光致断裂基团。同时,化学家们一直致力于发展生物相容性和光切割效率更高的光敏基团, 在某些情况下可替代邻硝基苄基及其衍生物,在化学生物学研究中使用的光致断裂基团还有香豆素类衍生物、对羟基苯乙酰基等。

|
图 1 常用光敏感基团 Fig.1 Several caging groups used in photochemical biology |
光致异构基团则是一类在不同波长下发生可逆异构化的光敏基团,包括在光照下发生可逆顺反异构的偶氮苯、二芳基乙烯和可逆开关环的吡螺酮等。其中,偶氮苯由于便于合成、光异构化量子产率高、光异构化速度快等诸多优势,被广泛应用于材料科学和化学生物学研究中[7]。光异构基团能够可逆地调控生物过程,提供光致断裂基团无法实现的操控模式,在具体生物学问题的研究中可与光致断裂策略互为补充。
2 光敏感核苷酸核苷酸不仅是DNA/RNA细胞内生物合成的原料,也在各种关键生命过程中扮演不同的重要角色,如代谢过程和离子转运的能量来源(ATP,GTP)、激活信号通路的信使分子(cAMP、cGMP、ADPR和cADPR等)、催化酶反应的辅酶(NAD、NADP)等。对核苷酸及其类似物的特殊位点进行光敏感基团的结构修饰,能够暂时封闭其功能,使相关生物过程无法进行。而在特定的时间点或位置施加光照,切除光敏基团后,可恢复其活性,触发生物学事件,因而可以用于生物体内快速和动态生命过程的研究。对核苷酸及其类似物的光敏修饰策略包括在碱基、糖环的羟基及磷酸基骨架上的光敏感基团修饰[8](图 2)。
光敏感ATP作为第一个合成的光敏生物活性分子,如今已成为商品化试剂用于生物学研究(图 3)。由于ATP是生物体的主要能量来源,细胞中许多蛋白具有ATP酶活性,只有在与ATP结合并将其水解释放能量之后,才能产生作用。而光敏ATP不能被ATP酶水解,在光照除去光敏基团后才可使其恢复成正常的底物ATP,为需要能量的动态生命过程的研究提供了新的研究手段。动力蛋白、肌球蛋白和肌动蛋白是一类具有ATP酶活性的马达蛋白,在细胞骨架形成、有丝分裂和细胞运动等重要生命过程中发挥作用[9]。Higuchi等[10]将光敏感ATP用于测量动力蛋白在微管上移动时力的产生模式;Chaen等[11]用光敏感ATP在光照激发后置换Cy3标记的ATP,证明了肌动蛋白在肌球蛋白上向两极滑动的不同速率,取决于水解ATP生成ADP的释放速率。

|
图 3 常用光敏ATP及其在生物学研究中的应用 Fig.3 Caged ATP and its applications in biological research |
另一类经典的ATP酶则是无处不在的离子泵,如钠钾泵、钙离子泵,以及细菌独有的钾离子转运系统KdpFABC等。离子转运是一个高度动态且快速的过程,光敏感ATP在离子转运的机制研究中有特别的优势。Kaplan等[4]最早合成的光敏感ATP也是用于研究红细胞膜上的钠钾泵。近来,Apell等[12]利用光敏感ATP详细研究了KdABC蛋白的钾离子转运机制,他们用紫外光照射光敏ATP释放正常的ATP底物,从而触发KdpFABC的磷酸化。同时,用电致发光染料检测KdpFABC膜结构域的电荷转移,证明钾离子是在蛋白磷酸化和变构后方与其膜结构域结合,对KdpFABC转运的Post-Albers模型进行了补充。另外,结合结构生物学手段,科学家们还能够像拍电影一样直观地“看到”大分子的运动过程。如Sasaki等[13]利用光敏ATP对伴侣蛋白进行紫外线激发的衍射X射线跟踪(DXT)研究,揭示了伴侣蛋白转动和闭合的动态过程等。
鸟嘌呤三磷酸(GTP)与G蛋白结合(Ras、Rho、Rab等)诱导其构象发生改变,在G蛋白偶联受体(GPCR)介导的各种跨膜信号转导过程中发挥重要作用。光敏感GTP亦被用于GPCR介导的信号通路分子层面上的精细研究。Somlyo等[14]将光敏感GTP用于研究鸟嘌呤核苷交换因子RhoGEF与GPCR介导的钙敏化效应的关系。平滑肌的收缩包括两个事件:1) 由激动剂与GPCR结合引起G结构域构象改变,激活下游通路导致钙电流产生;2) RhoA/ROCK通路刺激的钙敏化。已知G结构域能够激活RhoGEF,并且RhoGEF对钙敏化有调节作用,但对具体的过程尚不清楚。在这项研究中,研究者在利用RhoGEF敲除模型验证了RhoEGF在钙敏化中的作用后,用光敏感去氧肾上腺素(GPCR激动剂)、光敏感GTP和光敏感GTP/RhoA复合物,对信号通路的动态过程进行研究,测量了从光解释放活性物质到收缩力产生的时间,从而证明RhoGEF的募集和激活是钙电流产生与钙敏化之间延迟的主要原因。
环磷酸腺苷(cNMP)是另一类作为第二信使在细胞信号通路中发挥作用的特殊核苷酸。依赖于cAMP和cGMP的环磷酸腺苷门控离子通道不仅在正常生理活动中具有重大意义,其功能失常也与各种疾病息息相关。已报道的不同光敏基团修饰的光敏cNMP有20多种[8],这些光敏cNMP被广泛用于研究各种cNMP依赖的生物学过程。精子对卵细胞的趋化性是精子得以完成受精活动的重要性质之一。以海胆为例,精子趋化性产生的本质是由于卵细胞释放趋化因子,与精子鞭毛内的鸟苷酸环化酶结合,刺激cGMP的快速生成,进而打开环磷酸腺苷门控的钾离子通道,导致细胞膜的超极化,从而进一步开启钙离子通道,控制精子的运动[15, 16]。对于海生动物来说,精子如何应对海水波动导致的趋化因子时空分布和浓度的改变做出反应是一个很有意义的科学问题。Kaupp等[16]将光敏感cGMP与精子孵育后,用闪光间歇性地照射释放活性cGMP,同时记录钙离子流和精子运动轨迹的变化。结果发现,第二次光照后在钙离子浓度正常上升之前有一个短暂的下降,同时精子运动轨迹的曲率半径也变小。而且随着光照强度增加,钙离子下降程度增加,轨迹曲率半径的变化也增大。精子在活化肽(一种趋化因子)梯度浓度的溶液中也出现了相似运动轨迹。据此,他们推测精子响应趋化因子浓度变化改变运动轨迹是通过cGMP调节钙离子流实现的。光敏感cAMP则有报道被用于研究小鼠嗅觉感受器Cl-电流随时间的变化[17]、鸟嘌呤核苷交换因子Epac1的迁移[18]和衣藻鞭毛的运动[19]等动态过程。然而在将光敏核苷酸和其他光敏小分子一起使用时,由于常用的光敏基团吸收光谱都有一定重叠,无法满足精度更高或对更加复杂的生物学问题的研究。Davies等[20]发展了一种新的香豆素衍生物光敏感基团7-二乙氨基香豆素(DEAC450),其最大可用光激活波长位于440~480 nm,与常用的硝基芳环类光敏感基团错开。他们合成了DEAC450修饰的光敏cAMP,与CDNI(4-羧甲氧基-5, 7-二硝基吲哚啉基)修饰的GABA一同用于纹状体中间神经元动作电位双向调节的研究中。这两个光敏小分子对光照的响应表现出了很好的正交性,提示DEAC450在复杂生物机制研究中的应用前景。
除了对核苷酸磷酸基团的修饰,核苷酸的糖环和碱基上也可以进行有效的光敏感基团的修饰,并且已经在核酸测序领域得到应用。下一代测序技术在过去10年中飞速发展,各种高通量测序方法的出现,使人们得以更加快速全面地掌握基因组信息用于研究和临床应用[21]。其中,边合成边测序(sequencing-by-synthesis, SBS)方法,包括焦磷酸测序和循环可逆终止测序(cyclic reversible termination,CRT),依赖于DNA聚合酶对核苷酸的识别和利用。通过在核苷酸3′位引入光敏感基团,或利用光敏感基团连接用于检测的荧光团来合理设计可逆终止剂,能够弥补这类测序方法的一些缺陷。
传统焦磷酸测序法主要的问题是无法识别同聚物而导致的漏读或错读。Ju等[22]在核苷酸3′位修饰邻硝基苄基合成了光激活的可逆终止剂,并用于焦磷酸测序中。由于3′羟基被封闭,一次只能添加并读取一个碱基,读取结束后,光照脱除修饰基团重新暴露3′位羟基,继续合成和测序,这种方法避免了焦磷酸测序法读取同聚物序列时一次添加多个碱基的问题,提高了测序的准确性。而CRT方法中,提高测序精度和增加读长的关键之一则是可逆终止剂的优化,在设计中,一般要考虑的是减小修饰对聚合酶识别作用的影响和提高每次成像后修饰基团移除的速率和效率。由于3′位光敏修饰的核苷酸只能被少数聚合酶识别,Metzker等对碱基光敏修饰的核苷酸作为可逆终止剂的可能性进行了一系列探索。他们首先合成了N6位修饰邻硝基苄基的脱氧三磷酸腺苷单体[23],并证明这类修饰核苷酸是聚合酶的良好底物,然而较差的光脱除效率和普适性限制了其进一步应用。于是他们转而利用天然存在的5位羟甲基修饰的脱氧胸腺嘧啶核苷三磷酸(HOMedNTP)[24-26],合成了一套4种邻硝基苄基修饰的HOMedNTP(图 4)。同时,验证了聚合酶对这些核苷高效的利用率和良好的选择性,并利用叔丁基控制苄基α位的构象提高了光敏基团的切除速率。这套光敏感核苷三磷酸用于CRT测序中有望解决原有方法中存在的问题。
3 光敏感寡聚核苷酸 3.1 光敏感反义寡聚核苷酸反义寡聚核苷酸通过与靶mRNA特异性杂交阻断或抑制基因的表达,广泛用于研究靶基因和核酸药物的开发。为反义寡聚核苷酸装上光学“开关”,就能在特定时间点或区域通过光照恢复其活性,实现对基因表达时间或空间上的精确调控,这将有助于对复杂的基因表达模式及基因网络的研究。
构建光敏感反义寡聚核苷酸的一种策略是在寡聚核苷酸的磷酸骨架或碱基引入多个光敏感基团[27](图 5)。两者均通过干扰反义寡聚核苷酸与靶RNA的杂交或抑制后续核酸酶的结合,发挥对其活性的封闭作用。然而相较于磷酸骨架的修饰,碱基的修饰由于直接干扰了配对氢键的形成,功能封闭的效果较好,因而应用更为普遍。Deiters等[28]首先在反义硫代寡聚核苷酸引入3~4个6-硝基-3, 4-亚甲二氧苄氧甲基(NPOM)修饰的胸腺嘧啶核苷,光照后实现对哺乳动物细胞中荧光素酶基因敲除的空间调控。该策略亦被应用于吗啡啉寡聚核苷酸对斑马鱼和非洲爪蟾蜍胚胎中的EGFP及内源的chordin基因进行光控表达[29]。另外,通过在microRNA的反义寡聚核苷酸antagomir中插入NPOM修饰的尿嘧啶核苷[30],光敏感antagomir亦获得对microRNA功能的光调控能力,因而在microRNA功能和网络研究中有潜在的应用价值。

|
图 5 3种反义寡核苷酸光敏修饰策略 Fig.5 Three caging strategies for antisense oligonucleotides |
然而,引入多个光敏感基团,不仅不易将其完全脱除,还增加了设计和筛选的难度。针对这些问题,科学家们发展了一系列利用光敏感连接臂连接一段抑制性的互补正义链,形成发夹类似结构,从而暂时封闭反义寡聚核苷酸活性的方法(图 5)。在这种方法中,断裂连接臂释放反义寡聚核苷酸发挥作用只需要切割一个光敏感基团,一定程度上解决了多个碱基光敏感修饰方法的光控效果不理想的问题。利用这种策略设计的光敏感反义寡聚核苷酸,已成功应用于硫代磷酸寡聚核苷酸在白血病细胞中对cmyb基因的光调控[31],肽核酸和吗啡啉寡聚核苷酸在斑马鱼胚胎中对chordin和β-catenin等内源基因表达的光调控[32-34]。基于类似策略,Tang等[35]又设计了用偶氮苯作为连接臂,在反义寡聚核苷酸的两端分别连接一段抑制互补链,形成哑铃状结构的光敏感反义寡聚核苷酸,并将其用于RNase H对mRNA体外切割的可逆光调控。通过改变互补链和反义链的长度筛选出了光调控效果最好的组合,为后续细胞实验的进行奠定基础。发夹结构的设计虽然一定程度上解决了引入多个光敏感基团存在的问题,但是由于受细胞环境的复杂性,以及互补链与靶mRNA对反义寡聚核苷酸竞争结合的动态平衡等因素的影响,体外筛选出的光敏感反义寡聚核苷酸在溶液中的RNA降解和细胞内基因沉默功能的光调控效果并不一致,从而增加了筛选的难度和不确定性[34]。
为了克服以上策略的不足,Tang和Chen等[35-38]设计了一类环状光敏感寡聚核苷酸(图 5)。通过将反义寡聚核苷酸两端用光敏感连接臂连接成环,利用环状的二级结构实现对反义寡聚核苷酸活性的暂时沉默。这种策略避免了正义链和光敏感基团插入位置等设计筛选的繁琐过程,且具有良好的光调控效果。Tang等将此策略用于RNase H对RNA酶解的光调控,并探究了不同大小环状光敏反义寡聚核苷酸的光控效果,结果发现20 mer的环状反义寡聚核苷酸在光照前不与靶mRNA结合,光照后则很好地恢复结合活性[35]。在此基础上,他们又设计了环状硫代2′-OMeRNA反义核酸[37]和光敏感反义吗啡啉寡聚核苷酸[36],并分别用于对细胞内外源基因GFP表达的光调控、内源基因β-catenin和ntl在斑马鱼胚胎中功能的光调控。而Chen课题组则对这种策略的应用进行了拓展[38]。他们使用不同波长响应的光活性基团合成连接臂,设计合成了波长选择性激发的光敏感环状吗啉寡聚核苷酸。筛选出在体内实验中效果较好的邻硝基苄基(NB)和二乙胺基香豆素(DEACM)连接臂的环状吗啡啉寡聚核苷酸后(图 6),他们用其对斑马鱼胚层图式的形成机制进行了研究。Spt和flh基因在斑马鱼中胚层图式形成中发挥主要作用,spt突变导致躯干体节形成异常,flh则能够抑制spt在轴中胚层的异位表达。同时将合成的靶向spt的NB环状吗啉寡聚核苷酸和靶向flh的DEACM环状寡聚核苷酸一同注射入斑马鱼鱼卵的胚胎细胞中。在注射后3.5 h和5~7 h分别用405或470 nm和365 nm的光照射胚胎,相继沉默了flh和spt基因,验证了异位表达的spt在flh敲除的胚胎中诱导轴向发育的时间点。

|
图 6 对基因表达波长选择性的光调控[38] Fig.6 Sequential gene silencing using wavelength-selective caged oligonucleotides[38] |
RNA干扰及CRISPR/cas9系统是一类从生物体内发现,并被广泛应用的基因研究工具。RNA干扰的原理为通过Dicer等核酸内切酶对dsRNA前体进行切割处理形成siRNA,siRNA的反义链与细胞内的酶组装成RNA诱导的沉默复合体(RISC),对靶mRNA进行切割导致基因沉默。对siRNA进行光敏感基团修饰的努力集中在对RISC的形成及活性进行干预。Friedman等[39]最早通过随机标记策略在siRNA的磷酸骨架上引入1-(4, 5-二甲氧基-2-硝基苯基)乙烷基(DMNPE)实现对siRNA活性的光调控。这种方法同样存在光敏感基团太少则封闭不完全,太多则难以完全恢复siRNA活性的问题[40],因而更多的研究转向对siRNA发挥活性影响较大的位点进行精准修饰。
siRNA反义链5′末端的识别在RISC的组装中有重要作用[41],Nguyen等通过在5′末端的磷酸基缀连1-(2-硝苄基)-乙烷基(NPE)连接的生物素分子[42]对靶向甘油醛-3-磷酸脱氢酶(GAPDH)基因的siRNA,实现了70%~80%活性的沉默。然而在5′端修饰的策略中,研究者们普遍观察到siRNA活性封闭不完全的现象[42, 43]。Jain等[44]推测这种现象的原因可能是光活性基团位阻不够大, 或其在细胞内被相关酶切除,据此, 他们将环十二烷基与DMNPE连接,设计了一种新的大位阻光活性基团,并将其修饰在dsRNA的所有4个末端,阻碍了Dicer对dsRNA的加工,实现光照前对siRNA活性的完全封闭,而光照切除光活性基团后dsRNA被Dicer酶正常识别加工,组装成RISC恢复活性。这种策略可以达到较好的光控效果,但是大位阻基团的潜在体内毒性可能限制其进一步的应用。2016年,Tang等[45]发展了在siRNA反义链的5′末端修饰单个维生素E标记的光敏感siRNA,实现对荧光素酶和绿色荧光蛋白基因在细胞内高达18.6倍的光调控效果。作者期望通过缀连维生素E增加脂溶性和靶向性,提高siRNA的递送效率,却在实验中意外观察到siRNA正义链5′端维生素E修饰也有很好的抑制siRNA活性的效果(图 7),这与之前的文献报道相违。进一步的siRNA追踪实验证明, siRNA的沉默不是主要因维生素E自身的位阻效应,而是由于维生素E结合蛋白与维生素E结合,极大地增加了siRNA末端的位阻效应,阻止RISC复合物的形成和后续的siRNA活化。
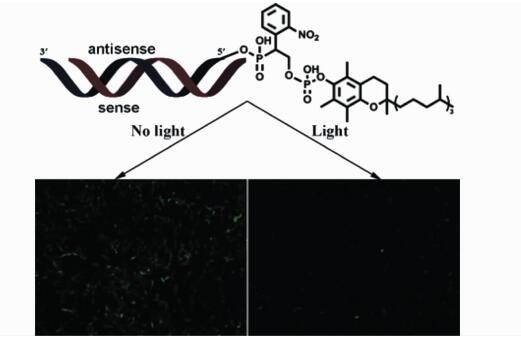
|
图 7 维生素E单标记的光敏感siRNA及其光调控效果[45] Fig.7 Photochemical regulation of gene expression using caged siRNAs with single terminal vitamin E modification[45] |
RISC形成后,要执行基因沉默功能,还需经过siRNA介导的对靶mRNA的识别和切割。Heckel等[46]将上游接近切割位点(距5′端10-11nt)的1~2个核苷替换为碱基修饰有2-(2-硝苄基)丙基(NPP)的脱氧核糖核苷,使修饰的RNA丧失了至少90%的活性。替换2个核苷的光封闭效果更好,但光照后活性无法完全恢复。Deiters等[47]后续合成鸟嘌呤6位和尿嘧啶亚胺N上修饰有邻硝基亚甲二氧苯甲氧甲基(NPOM)的核苷亚磷酰胺单体,并将其定点插入切割区域附近和负责识别靶mRNA的种子区域中。他们利用这种策略解决了光照前siRNA活性泄漏和光照后活性恢复差的问题,并且首次将光敏siRNA用于内源基因的光诱导沉默。
限制光敏siRNA的应用的关键因素,是激发常用光敏基团的紫外光线的低穿透性,这也是光敏技术在生物学研究和临床研究中应用的重要障碍之一。为解决这一问题,研究者们进行了很多尝试[48-50]。Meyer等[51]设计了一种单重态氧介导可见光激发的光敏siRNA系统,如图 8。siRNA反义链5′末端连接一个对1O2敏感的9-烷氧蒽,并通过阻止5′端被磷酸化封闭来siRNA的活性。而正义链3′端连接不同吸收波长的光敏剂(P1和P2吸收红光,P3吸收绿光),光敏剂被可见光激发,产生单重态氧切割5′端的修饰基团,使siRNA活性得以恢复。利用这套系统,他们用波长长于600 nm、无细胞毒性的光照成功实现了在细胞中激活siRNA。
CRISPR/Cas9系统自被发现以来发展迅猛,俨然已超越siRNA、shRNA等,成为目前使用最广的基因编辑工具,无论是在生物学还是临床研究中,都有极大的应用潜力。而设计光激活的CRISPR无疑能够大大拓展其应用范围。光敏CRISPR的设计策略之一是对Cas9酶进行改造,包括:利用基因密码子扩展技术定点插入光活性基团[52],连接光敏感的蛋白结构域[53]等。最近,Jain等[54]率先将光断裂寡聚核苷酸链的策略用到光敏sgRNA的设计中。如图 9所示,他们设计了一系列不同长度,位于sgRNA靶向区域不同位置,并且包含光活性基团的互补ssDNA作为sgRNA的保护序列。当保护序列与sgRNA杂交,形成的复合物具有高的Tm值,较为稳定,能够阻止sgRNA与靶序列结合发挥作用。光照后,保护序列断裂成小片段,Tm值降低,释放sgRNA实现靶向切割。作者还对保护序列的骨架,光敏感基团间隔的序列长度及寡聚核苷酸3′末端进行了改进与修饰的尝试,发现2′-OMe RNA骨架亦能发挥与ssDNA相似的保护效果。在细胞实验中,光活性基团间隔8个核苷的序列设计降低了光控效果,这可能是因为光照后形成的小片段仍保留结合sgRNA的能力。此外,ssDNA的3′端对FAM修饰表现出良好的耐受性,这为sgRNA的间接标记提供了可能,或可用于成像和机制研究。目前报道的sgRNA保护的光敏CRISPR系统可实现靶向多基因能力。相对于改造Cas9酶,对sgRNA的修饰、功能化和表征更为方便,利于进一步的优化和推广应用。未来的研究方向应集中于提高现有系统的光控效果和特异性,引入不同波长的正交的光敏感基团,实现多基因编辑的时空调控,以及将光敏寡聚核苷酸不同的设计策略应用于sgRNA以克服现有系统的不足等几个方面。

|
图 9 CRISPR设计原理及基因编辑的光调控效果[54] Fig.9 Concept of light-activated CRISPR and its photomodulating of gene expression[54] |
随着研究的深入,人们发现核酸不仅在复制、转录和翻译过程中发挥调控功能,也通过直接与蛋白相互作用及自身的核酶活性在生命过程中发挥作用。核酸工具的开发思路也大大拓展,许多对蛋白质,包括酶、受体等直接调节以及具有酶活性的核酸工具被开发出来,如脱氧核酶、核酸适配体等。核酶和脱氧核酶是一类具有催化活性(主要催化目标RNA的剪切)的寡聚核苷酸,在生物传感器、药物治疗和功能基因组学研究中均扮演了重要的角色。核酶及脱氧核酶的光敏修饰集中在催化中心和结合臂,包括引入碱基修饰的核苷[55-57],或光致断裂的连接臂[58]。核酸适配体是通过SELEX技术体外筛选获得的具有特异性识别能力的寡聚核苷酸,其与受体高亲和力结合后能够诱导构象改变[59], 发挥调节功能。Mayer和Heckel等在凝血酶适配体的序列中插入NPE和NPP修饰的碱基,通过阻碍适配体和受体结合或影响适配体的折叠结构,来调节其功能[60, 61]。相应的工作已经有较多的综述进行了详细的阐述[1, 5]。
在核酶、脱氧核酶和核酸适配体的光敏修饰策略中,插入碱基的方法存在光敏感单体合成复杂的问题。近年,一些通过合成后定点修饰的策略对核酸功能进行光调控的方法有一定的发展。Xiang等[62]利用二乙基硫代磷酸盐与2-溴-4′-对羟基苯乙酮反应生成TFFP-OH的反应,在核酸硫代磷酸骨架上引入光敏感基团。由于通过固相合成能对核酸任意位点的磷酸进行硫代,这种方法可以实现对磷酸骨架的定点修饰。他们已将这种方法用于8~17和10~23核酶对目标RNA的切割活性[62]、G-四链体脱氧核酶的过氧化物酶活性以及G-四链体适配体结合血红素[63]的光调控。
Zhou等[64]利用光异构分子偶氮苯的衍生物通过光异构化实现核酸适配体对酶活性的可逆光控调节(图 10)。Zhou课题组[65]在前期工作中证实了Razo不同构象对人端粒DNA G-四链体形成的影响。在此基础上,他们在端粒DNA的5′端和3′端连接两个不同的凝血酶适配体29Apt和15Apt。当Razo为顺式构象时,端粒DNA无法形成四链体而处于伸展状态,两个核酸适配体可与凝血酶结合抑制其活性。用波长大于400 nm的可见光照射时,Razo转化为反式结构,诱导G四链体的形成,将两个适配体拉近,使其无法与凝血酶结合,凝血酶的活性恢复。这是首次用光活性小分子通过非修饰的方式对适配体的功能进行光控调节。Baigl等[66]亦合成了一系列胍基取代的偶氮苯衍生物,用于光控DNA双链的形成。这类光活性小分子未来可进一步应用到核酸化学生物学研究中。

|
图 10 光活性小分子对核酸适配体活性的调节[64] Fig.10 Light-regulation of aptamer using small molecular photo-switch[64] |
最近,Heckel等[67]将环状寡聚核苷酸的设计用到长链(90nt)的C10适配体中。如何提高长链核酸的光控效果,是领域内一直未能解决的问题。反义寡聚核苷酸的环化策略虽然有诸多优点,但将其应用于长链核苷酸时会因高的背景活性导致光控效果减弱。Heckel等[67]采用一种新的环化策略(图 11),合成了O6修饰邻硝基苄基或香豆素炔基衍生物(NPBY和DEACBY)的脱氧胸腺嘧啶核苷单体,将其插入寡聚核苷酸的序列中。利用1, 4-二(叠氮甲基)苯作为连接臂,通过铜催化叠氮炔环加成(CuAAC)的点击化学反应连接插入修饰碱基的两端来实现成环。这种方法可以通过改变插入修饰碱基的位置来控制环的大小。他们进一步将修饰碱基的核苷分别插入C10的7、37、73位,并用1, 3, 5-三(叠氮甲基)作为连接臂,构建了一个双环的束缚结构,这种结构使C10无法形成与伯基特氏淋巴瘤细胞表面受体结合所需要的G-四链体。光照后,C10恢复线性并可以形成其原有的二级结构而被细胞特异性摄取。这种方法能够普适性地用于长链核苷酸,包括任意序列的适配体和一些核酶的光照调控中。
另一方面,Toll样受体家族对外源核酸的识别和免疫响应,则是体内核酸直接与蛋白相互作用触发生物学事件的典型例子[68]。如果能用光控制免疫反应,将有助于解答免疫系统如何通过受体激活识别感染等生物学问题,在免疫相关疾病中也有潜在应用价值。CpG作为一种免疫佐剂通过结合Toll样受体9(TLR9) 刺激免疫反应。Govan等[69]设计合成了两种光敏CpG寡聚核苷酸:将4个NPOM修饰的胸腺嘧啶核苷均匀地插入序列中(CpG-4A)或集中在3′或5′末端(CpG-4B),并检验了其对TLR 9介导的免疫反应的光控效果。CpG-4A在光照后仍然无法激活TLR9,刺激IL-6的分泌,而CpG-4B也只能恢复部分活性。免疫相关机制是目前疾病研究的热点之一,目前针对核酸及其识别受体相互作用的光调控报道较少,这或许是今后的方向之一。
4 光敏DNA/RNA实现基因表达调控的另一种方法是直接利用光敏感基团对DNA质粒或mRNA进行暂时的活性封闭。早期的工作主要通过将质粒DNA[70]或mRNA[71]与光敏感小分子混合,在核酸磷酸骨架上随机插入多个光敏感基团,从而实现基因的暂时沉默。然而由于插入的光敏感基团数量过多,要实现它们的完全切除,对光的强度和光照时间均有较高要求,这种方法在光照后只能实现部分的活性恢复。更加有效的策略则是在对转录、翻译有特殊作用的区域进行定点的光敏感修饰。
Liang等[72]将偶氮苯共价连接于T7启动子,利用PCR和酶法连接结合的方法,构建了光响应的质粒,实现对GFP体外表达的可逆光调控。Deiters等[73]将光敏感基团修饰碱基的策略应用到光响应质粒的构建中(图 12)。他们在EGFP质粒的TATA box两侧插入了NT.BstNB的酶切位点,使包含TATA box的小片段能通过酶切除,并通过酶法连接将其替换成一段人工合成的,并在TATA box定点插入光敏感基团NPOM修饰胸腺嘧啶核苷的寡聚核苷酸。NOPM的修饰阻碍了转录因子与TATA box的结合,从而抑制了基因的表达。利用这个质粒体系,他们成功实现了对细胞和斑马鱼中外源荧光报告基因表达的光调控,及光诱导的内源信号蛋白过表达。
5 总结与展望光是一种非常方便和非侵入式的刺激手段。光激活技术已经成为一种非常高效的化学生物学研究工具,广泛应用于激活生物活性分子(特别是核酸生物分子)的生物功能,并在分子、细胞和某些模式动物水平上取得了非常大的成功。通过选择合适的光敏感基团(邻硝基苄基、对羟基苯乙酰基、香豆素和偶氮苯及其衍生物等)的组合,生物分子可能实现不同波长的顺序激活,以及在时间、空间,甚至浓度上的功能调控。另一方面,不同种类的功能核酸可能也需要采用不同的光敏感修饰策略来实行对核酸功能调控的优化。与此同时,光激活技术由于受到光照波长的限制(目前主要在紫外光区和部分蓝光区),目前只能在细胞水平和某些模式动物水平(如透明的斑马鱼)作为研究细胞生命过程和特定基因调控的研究工具。未来的光化学生物学研究将需要着眼于更高等生物体系(如小鼠模型)的研究。具有更好穿透力的红光激活核酸的功能将是核酸光化学生物学需要解决的关键技术问题,将为生物体系中的核酸药物的研究提供助力。
| [1] | Liu Q, Deiters A. Optochemical control of deoxyoligonucleotide function via a nucleobase-caging approach[J]. Accounts of Chemical Research, 2013, 47(1): 45–55. |
| [2] | Brieke C, Rohrbach F, Mayer G, Heckel A. Light-controlled tools[J]. Angewandte Chemie International Edition, 2012, 51(34): 8446–8476. DOI:10.1002/anie.201202134 |
| [3] | Szymański W, Beierle J M, Kistemaker H A, Mayer G, Heckel A. Reversible photocontrol of biological systems by the incorporation of molecular photoswitches[J]. Chemical Reviews, 2013, 113(8): 6114–6178. DOI:10.1021/cr300179f |
| [4] | Kaplan J H, Forbush B, Hoffman J F. Rapid photolytic release of adenosine 5'-triphosphate from a protected analogue: utilization by the Na: K pump of human red blood cell ghosts[J]. Biochemistry, 1978, 17(10): 1929–1935. DOI:10.1021/bi00603a020 |
| [5] | Tang X. Photochemical biology of caged nucleic acids[J]. Photochemistry, 2013, 41: 319–341. DOI:10.1039/1465-1947 |
| [6] | Klán P, Šolomek T, Bochet C G, Blanc A, Givens R, Rubina M, Popik V, Kostikov A, Wirz J. Photoremovable protecting groups in chemistry and biology: reaction mechanisms and efficacy[J]. Chemical Reviews, 2013, 113(1): 119–191. DOI:10.1021/cr300177k |
| [7] | Szymański W, Beierle J M, Kistemaker H A, Velema W A, Feringa B L. Reversible photocontrol of biological systems by the incorporation of molecular photoswitches[J]. Chemical Reviews, 2013, 113(8): 6114–6178. DOI:10.1021/cr300179f |
| [8] | Tang X, Zhang J, Sun J, Wang Y, Wu J, Zhang L. Caged nucleotides/nucleosides and their photochemical biology[J]. Organic & Biomolecular Chemistry, 2013, 11(45): 7814–7824. |
| [9] | Vale R D. The molecular motor toolbox for intracellular transport[J]. Cell, 2003, 112(4): 467–480. DOI:10.1016/S0092-8674(03)00111-9 |
| [10] | Shingyoji C, Nakano I, Inoue Y, Higuchi H. Dynein arms are strain-dependent direction-switching force generators[J]. Cytoskeleton, 2015, 72(8): 388–401. DOI:10.1002/cm.v72.8 |
| [11] | Maruta T, Kobatake T, Okubo H, Chaen S. Single turnovers of fluorescent ATP bound to bipolar myosin filament during actin filaments sliding[J]. Biophysics, 2013, 9: 13–20. DOI:10.2142/biophysics.9.13 |
| [12] | Damnjanovic B, Apell H J. Role of protons in the pump cycle of KdpFABC investigated by time-resolved kinetic experiments[J]. Biochemistry, 2014, 53(19): 3218–3228. DOI:10.1021/bi500336w |
| [13] | Sekiguchi H, Nakagawa A, Moriya K, Makabe K, Ichiyanagi K, Nozawa S, Sato T, Adchi S, Kuwajima K, Yohda M, Sasaki Y C. ATP dependent rotational motion of group Ⅱ chaperonin observed by X-ray single molecule tracking[J]. Plos One, 2013, 8(5): e64176. DOI:10.1371/journal.pone.0064176 |
| [14] | Artamonov M V, Momotani K, Stevenson A, Trentham D R, Derewenda U, Derewenda U, Derewenda Z S, Read P W, Gutkind J S. Agonist-induced Ca2+, sensitization in smooth muscle[J]. Journal of Biological Chemistry, 2013, 288: 34030–34040. DOI:10.1074/jbc.M113.514596 |
| [15] | Bönigk W, Loogen A, Seifert R. An atypical CNG channel activated by a single cGMP molecule controls sperm chemotaxis[J]. Science Signaling, 2009, 2(94): ra68. |
| [16] | Kashikar N D, Alvarez L, Seifert R, Gregor I, Jackie O, Beyermann M, Krause E, Kaupp U B. Temporal sampling, resetting, and adaptation orchestrate gradient sensing in sperm[J]. Journal of Cell Biology, 2012, 198(6): 1075–1091. DOI:10.1083/jcb.201204024 |
| [17] | Boccaccio A, Menini A. Temporal development of cyclic nucleotide-gated and Ca2+ -activated Cl- currents in isolated mouse olfactory sensory neurons[J]. Journal of Neurophysiology, 2007, 98(1): 153–160. DOI:10.1152/jn.00270.2007 |
| [18] | Ponsioen B, Gloerich M, Ritsma L, Rehmann H, Bos J L, Jalink K. Direct spatial control of epac1 by cyclic AMP[J]. Molecular & Cellular Biology, 2009, 29(10): 2521–2531. |
| [19] | Saegusa Y, Yoshimura K. cAMP controls the balance of the propulsive forces generated by the two flagella of chlamydomonas[J]. Cytoskeleton, 2015, 72(8): 412–421. DOI:10.1002/cm.v72.8 |
| [20] | Olson J P, Banghart M R, Sabatini B L, Ellis-Davies G C. Spectral evolution of a photochemical protecting group for orthogonal two-color uncaging with visible light[J]. Journal of the American Chemical Society, 2013, 135(42): 15948–15954. DOI:10.1021/ja408225k |
| [21] | Goodwin S, Mcpherson J D, Mccombie W R. Coming of age: ten years of next-generation sequencing technologies[J]. Nature Reviews Genetics, 2016, 17(6): 333–351. DOI:10.1038/nrg.2016.49 |
| [22] | Wu J, Zhang S, Meng Q, Cao H, Li Z, Li X, Shi S, Kim D H, Bi L, Turro N J, Ju J. 3'-O-modified nucleotides as reversible terminators for pyrosequencing[J]. Proceedings of the National Academy of Sciences, 2007, 104(42): 16462–16467. DOI:10.1073/pnas.0707495104 |
| [23] | Wu W, Stupi B P, Litosh V A, Mansouri D, Farley D, Morris S, Metzker S, Metzker M L. Termination of DNA synthesis by N6 -alkylated, not 3'-O-alkylated, photocleavable 2'-deoxyadenosine triphosphates[J]. Nucleic Acids Research, 2007, 35(19): 6339–6349. DOI:10.1093/nar/gkm689 |
| [24] | Litosh V A, Wu W, Stupi B P, et al. Improved nucleotide selectivity and termination of 3'-OH unblocked reversible terminators by molecular tuning of 2-nitrobenzyl alkylated HOMedU triphosphates[J]. Nucleic Acids Research, 2011, 39(6): e39. DOI:10.1093/nar/gkq1293 |
| [25] | Stupi B P, Li H, Wang J, Wu W, Morris S E, Litosh V A, Muniz J, Hersh M N, Metzker M L. Stereochemistry of benzylic carbon substitution coupled with ring modification of 2-nitrobenzyl groups as key determinants for fast-cleaving reversible terminators[J]. Angewandte Chemie International Edition, 2012, 51(7): 1724–1727. DOI:10.1002/anie.v51.7 |
| [26] | Gardner A F, Wang J, Wu W, Karouby J, Li H, Stupi B P, Jack W E, Hersh M N, Metzker M L. Rapid incorporation kinetics and improved fidelity of a novel class of 3'-OH unblocked reversible terminators[J]. Nucleic Acids Research, 2012, 40(15): 7404–7415. DOI:10.1093/nar/gks330 |
| [27] | Ghosn B, Haselton F R, Gee K R, Monroe W T. Control of DNA hybridization with photocleavable adducts[J]. Photochemistry & Photobiology, 2009, 81(4): 953–959. |
| [28] | Young D D, Lusic H, Lively M O, Yoder J A, Deiters A. Gene silencing in mammalian cells with light-activated antisense agents[J]. Chembiochem, 2008, 9(18): 2937–2940. DOI:10.1002/cbic.v9:18 |
| [29] | Deiters A, Garner R A, Lusic H, Govan J M, Dush M, Nascone-Yoder N M, Yoder J A. Photocagedmorpholino oligomers for the light-regulation of gene function in zebrafish and, xenopus embryos[J]. Journal of the American Chemical Society, 2010, 132(44): 15644–15650. DOI:10.1021/ja1053863 |
| [30] | Connelly C M, Uprety R, Hemphill J, Deiters A. Spatiotemporal control of microRNA function using light-activated antagomirs[J]. Molecular Biosystems, 2012, 8(11): 2987–2993. DOI:10.1039/c2mb25175b |
| [31] | Tang X, Swaminathan J, Gewirtz A M, Dmochowski I J. Regulating gene expression in human leukemia cells using light-activated oligodeoxynucleotides[J]. Nucleic Acids Research, 2008, 36(2): 559–569. |
| [32] | Tang X, Maegawa S, Weinberg E S, Dmochowski I J. Regulating gene expression in zebrafish embryos using light-activated, negatively charged peptide nucleic acids[J]. Journal of the American Chemical Society, 2007, 129(36): 11000–11001. DOI:10.1021/ja073723s |
| [33] | Shestopalov I A, Sinha S, Chen J K. Light-controlled gene silencing in zebrafish embryos[J]. Nature Chemical Biology, 2007, 3(10): 650–651. DOI:10.1038/nchembio.2007.30 |
| [34] | Ouyang X, Shestopalov I A, Sinha S, Zheng G, Pitt C L, Li W H, Olson A J, Chen J K. Versatile synthesis and rational design of caged morpholinos[J]. Journal of the American Chemical Society, 2009, 131(37): 13255–13269. DOI:10.1021/ja809933h |
| [35] | Tang X, Su M, Yu L, Lv C, Wang J, Li Z. Photomodulating RNA cleavage using photolabile circular antisense oligodeoxynucleotides[J]. Nucleic Acids Research, 2010, 38(11): 3848–3855. DOI:10.1093/nar/gkq079 |
| [36] | Wang Y, Wu L, Wang P, Lv C, Yang Z, Tang X. Manipulation of gene expression in zebrafish using caged circular morpholino oligomers[J]. Nucleic Acids Research, 2012, 40(21): 11155–11162. DOI:10.1093/nar/gks840 |
| [37] | Wu L, Wang Y, Wu J, Lv C, Wang J, Tang X. Caged circular antisense oligonucleotides for photomodulation of RNA digestion and gene expression in cells[J]. Nucleic Acids Research, 2013, 41(1): 677–686. DOI:10.1093/nar/gks996 |
| [38] | Yamazoe S, Liu Q, Mcquade L E, Deiters A, Chen J K. Sequential gene silencing using wavelength-selective caged morpholino oligonucleotides[J]. Angewandte Chemie International Edition, 2014, 53(38): 10114–10118. DOI:10.1002/anie.201405355 |
| [39] | Shah S, Rangarajan S, Friedman S H. Light-activated RNA interference[J]. Angewandte Chemie International Edition, 2005, 44(9): 1328–1332. DOI:10.1002/(ISSN)1521-3773 |
| [40] | Shah S, Jain P K, Kala A, Karunakaran D, Friedman S H. Light-activated RNA interference using double-stranded siRNA precursors modified using a remarkable regiospecificity ofdiazo-based photolabile groups[J]. Nucleic Acids Research, 2009, 37(13): 4508–4517. DOI:10.1093/nar/gkp415 |
| [41] | Chiu Y L, Rana T M. RNAi in Human Cells: Basic Structural and Functional Features of Small Interfering RNA[J]. Molecular Cell, 2002, 10(3): 549–561. DOI:10.1016/S1097-2765(02)00652-4 |
| [42] | Nguyen Q N, Chavli R V, Marques J T, Conrad P G, Wang D, He W, Belisle B E, Zhang A, Pastor L M, Witney F R, Morris M, Heitz F, Divita G, Williams B R, McMaster G K. Light controllable siRNAs regulate gene suppression and phenotypes in cells[J]. Biochimica Et Biophysica Acta Biomembranes, 2006, 1758(3): 394–403. DOI:10.1016/j.bbamem.2006.01.003 |
| [43] | Shah S, Friedman S H. Tolerance of RNA interference toward modifications of the 5' antisense phosphate of small interfering RNA[J]. Oligonucleotides, 2007, 17(1): 35–43. DOI:10.1089/oli.2006.0067 |
| [44] | Jain P K, Shah S, Friedman S H. Patterning of gene expression using new photolabile groups applied to light activated RNAi[J]. Journal of the American Chemical Society, 2010, 133(3): 440–446. |
| [45] | Ji Y, Yang J, Wu L, Yu L, Tang X. Photochemical regulation of gene expression using caged siRNAs with single terminal vitamin E modification[J]. Angewandte Chemie International Edition, 2016, 55(6): 2152–2156. DOI:10.1002/anie.201510921 |
| [46] | Mikat V, Heckel A. Light-dependent RNA interference with nucleobase-caged siRNAs[J]. RNA, 2007, 13(12): 2341–2347. DOI:10.1261/rna.753407 |
| [47] | Govan J M, Young D D, Lusic H, Liu Q, Lively M O, Deiters A. Optochemical control of RNA interference in mammalian cells[J]. Nucleic Acids Research, 2013, 41(22): 10518–10528. DOI:10.1093/nar/gkt806 |
| [48] | Braun G B, Pallaoro A, Wu G, Missirlis D, Zasadzinski J A, Tirrell M, Reich N O. Laser-activated gene silencing via gold nanoshell-siRNA conjugates[J]. ACS Nano, 2009, 3(7): 2007–2015. DOI:10.1021/nn900469q |
| [49] | Huang X, Hu Q, Braun G B, Pallaoro A, Morales D P, Zasadzinski J, Clegg D O, Reich N O. Light-activated RNA interference in human embryonic stem cells[J]. Biomaterials, 2015, 63: 70–79. DOI:10.1016/j.biomaterials.2015.06.006 |
| [50] | Yang Y, Liu F, Liu X, Xing B. NIR light controlled photorelease of siRNA and its targeted intracellular delivery based on upconversion nanoparticles[J]. Nanoscale, 2012, 5(1): 231–238. |
| [51] | Meyer A, Mokhir A. RNA interference controlled by light of variable wavelength[J]. Angewandte Chemie International Edition, 2014, 53(47): 12840–12843. DOI:10.1002/anie.201405885 |
| [52] | Hemphill J, Borchardt E K, Brown K, Asokan A, Deiters A. Optical control of CRISPR/Cas9 gene editing[J]. Journal of the American Chemical Society, 2015, 137(17): 5642–5645. DOI:10.1021/ja512664v |
| [53] | Nihongaki Y, Yamamoto S, Kawano F, Suzuki H, Sato M. CRISPR-Cas9-based photoactivatable transcription system[J]. Chemistry & Biology, 2015, 22(2): 169–174. |
| [54] | Jain P K, Ramanan V, Schepers A G, Dalvie N S, Panda A, Fleming H E, Bhatia. Development of light-activated CRISPR using guide RNAs with photocleavable protectors[J]. Angewandte Chemie International Edition, 2016, 55(40): 12628–12632. |
| [55] | Ting R, Lermer L, Perrin D M. Triggering DNAzymes with light: a photoactive C8 thioether-linked adenosine[J]. Journal of the American Chemical Society, 2004, 126(126): 12720–12721. |
| [56] | Lusic H, Young D D, Lively M O, Deiters A. Photochemical DNA activation[J]. Organic Letters, 2007, 9(10): 1903–1906. DOI:10.1021/ol070455u |
| [57] | Young D D, Lively M O, Deiters A. Activation and deactivation of DNAzyme and antisense function with light for the photochemical regulation of gene expression in mammalian cells[J]. Journal of the American Chemical Society, 2010, 132(17): 6183–6193. DOI:10.1021/ja100710j |
| [58] | Richards J L, Seward G K, Wang Y H, Dmochowski I J. Turning the 10-23 DNAzymeon and off with light[J]. Chembiochem, 2010, 11(3): 320–324. DOI:10.1002/cbic.v11:3 |
| [59] | Vinkenborg J L, Karnowski N, Famulok M. Aptamers for allosteric regulation[J]. Nature Chemical Biology, 2011, 7(8): 519–527. DOI:10.1038/nchembio.609 |
| [60] | Mayer G, Kröck L, Mikat V, Engeser M, Heckel. Light-induced formation of G-quadruplex DNA secondary structures[J]. Chembiochem, 2005, 6(11): 1966–1970. DOI:10.1002/cbic.v6:11 |
| [61] | Buff M C, Schäfer F, Wulffen B, Müller J, Pötzsch B, Heckel A, Mayer G. Dependence of aptamer activity on opposed terminal extensions: improvement of light-regulation efficiency[J]. Nucleic Acids Research, 2010, 38(6): 2111–2118. DOI:10.1093/nar/gkp1148 |
| [62] | Wang X, Feng M, Xiao L, Tong A, Xiang Y. Postsynthetic modification of DNA phosphodiester backbone for photocagedDNAzyme[J]. ACS Chemical Biology, 2016, 11(2): 444–451. DOI:10.1021/acschembio.5b00867 |
| [63] | Feng M, Ruan Z, Shang J, Xiao L, Tong A, Xiang Y. Photocaged G-quadruplexDNAzyme andaptamer by postsynthetic modification on phosphodiester backbone[J]. Bioconjugate Chemistry, 2016, 28(2): 549–555. |
| [64] | Tian T, Song Y, Wang J, Fu B, He Z, Xu X, Li A, Zhou X, Wang S, Zhou X. Small-molecule-triggered and light-controlled reversible regulation of enzymatic activity[J]. Journal of the American Chemical Society, 2016, 138(3): 955–961. DOI:10.1021/jacs.5b11532 |
| [65] | Wang X, Huang J, Zhou Y, Yan S, Weng X, Wu X, Deng M, Zhou X. Conformational switching of G-quadruplex DNA by photoregulation[J]. Angewandte Chemie International Edition, 2010, 49(31): 5305–5309. DOI:10.1002/anie.v49:31 |
| [66] | Bergen A, Rudiuk S, Morel M, Saux T L, Ihmeis H, Baigl D. Photodependent melting of unmodified DNA using a photosensitive intercalator: a new and generic tool for photoreversible assembly of DNA nanostructures at constant temperature[J]. Nano Letters, 2015, 16(1): 773–780. |
| [67] | Seyfried P, Eiden L, Grebenovsky N, Mayer G, Heckel A. Photo-tethers for the (multi-)cyclic, conformational caging of long oligonucleotides[J]. Angewandte Chemie, 2016, 56(1): 359–363. |
| [68] | Schlee M, Hartmann G. Discriminating self from non-self in nucleic acid sensing[J]. Nature Reviews Immunology, 2016, 16(9): 566–580. DOI:10.1038/nri.2016.78 |
| [69] | Govan J M, Young D D, Lively M O, Deiters A. Optically triggered immune response through photocaged oligonucleotides[J]. Tetrahedron Letters, 2015, 56(23): 3639–3642. DOI:10.1016/j.tetlet.2015.01.165 |
| [70] | Ando H, Furuta T, Tsien R Y, Okamoto H. Photo-mediated gene activation using caged RNA/DNA in zebrafish embryos[J]. Nature Genetics, 2001, 28(4): 317–325. DOI:10.1038/ng583 |
| [71] | Ando H, Furuta T, Okamoto H. Photo-mediated gene activation by using caged mRNA in zebrafish embryos[J]. Methods in Cell Biology, 2004, 77(77): 159–171. |
| [72] | Kamiya Y, Takagi T, Ooi H, Ito H, Liang X, Asanuma H. Synthetic gene involving azobenzene-tethered T7 promoter for the photocontrol of gene expression by visible light[J]. ACS Synthetic Biology, 2015, 4(4): 365–370. DOI:10.1021/sb5001092 |
| [73] | Hemphill J, Govan J, Uprety R, Tsang M, Deiters A. Site-specific promoter caging enables optochemical gene activation in cells and animals[J]. Journal of the American Chemical Society, 2014, 136(19): 7152–7158. DOI:10.1021/ja500327g |









