DNA是抗肿瘤药物的重要作用靶点。在正常细胞中存在着大量的双螺旋DNA,因此以双螺旋DNA为靶点的抗肿瘤药物容易产生严重的毒副作用[1, 2]。针对特定的核酸二级结构或核酸序列进行药物设计,合成毒性低、选择性更好的抗肿瘤药物是当今化学、生物、医药领域的一个重要发展趋势[3-5]。在人体的端粒末端含有TTAGGG重复碱基单元。细胞有丝分裂致使端粒d(TTAGGG)重复片段丢失,从而逐渐缩短[6],当端粒DNA长度缩短到一定程度时,染色体末端的功能就会遭到破坏,促使正常细胞逐步进入衰老和凋亡阶段[7-9]。但肿瘤细胞中端粒酶的逆转录作用能保持端粒的长度,使恶性肿瘤细胞获得无限增值能力[10, 11]。
研究发现人端粒d(TTAGGG)序列可通过Hoogsteen氢键作用形成四分体结构,并进一步经过π-π堆积形成G-四链体[12]。端粒酶的逆转录和表达均以单链DNA为模板;而G-四链体的形成改变了单链结构,从而抑制端粒酶的活性,可以诱导肿瘤细胞的凋亡[13, 14]。因此,能够诱导G四链体形成并稳定其结构的化合物具有潜在的靶向抗癌作用[15-19]。目前基于G-四链体结构的抗肿瘤药物Quarfloxin(CX-3543) 已经进入临床试验阶段并取得了良好的疗效[20]。
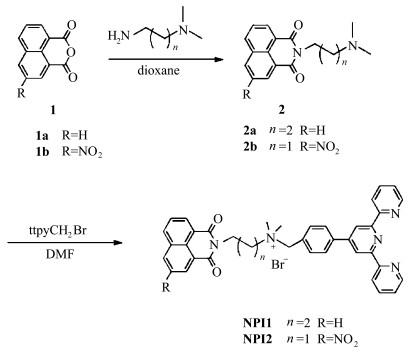
文献报道了许多能够稳定G-四链体的小分子化合物,如蒽醌类衍生物、苝类化合物、卟啉类化合物和吖啶衍生物等。这些化合物大多具有平面芳香大共轭体系且带有正电荷, 主要通过π-π堆积和静电作用两种结合模式与G-四链体发生作用[21, 22]。三联吡啶及其配合物具有大的π共轭结构,常用做G-四链体的稳定剂[23]。在三联吡啶的侧链引入正电荷可改善三联吡啶的水溶性和对DNA的亲和性,但是这些化合物对G-四链体DNA的选择性不高[24]。在三联吡啶配合物中引入具有抗癌活性的萘酰亚胺基团,可提高三联吡啶衍生物对G-四链体的选择性和抗癌活性[25]。本文将萘酰亚胺衍生物与三联吡啶通过共价连接,得到具有季铵盐结构的萘酰亚胺-三联吡啶衍生物(图 1),利用紫外吸收光谱、圆二色谱等研究了这些化合物与双链DNA和G-四链体DNA的相互作用,并采用AutoDock推测了化合物与G-四链体DNA的作用方式。
|
图 1 化合物NPI1和NPI2的合成路线 Fig.1 Synthesis scheme of NPI1 and NPI2 |
试剂:1, 8-萘二甲酸酐、N, N二甲基丙二胺、N, N二甲基乙二胺、4′-(对甲基苯基)-2, 2′:6′, 2″-三联吡啶、N, N-二甲基甲酰胺(DMF)、N-(2-羟乙基)哌嗪-N′-2-乙磺酸(HEPES)、小牛胸腺DNA(CT DNA)、4′-(对溴代亚甲基苯基)-2, 2′:6′, 2″-三联吡啶(ttpyCH2Br)均根据文献合成[26]。除非另有说明,光谱滴定实验都在10 mmol/L HEPES、pH 7.4、0.1 mol/L K+中进行。缓冲溶液为用二次蒸馏水配制的HEPES缓冲溶液(10 mmol/L,pH 7.4)。以下寡聚核苷酸由上海生工合成并经过纯化:Htelo(5′-GGG-TTA-GGG-TTA-GGG-TTA-GGG-3′)。
所用仪器:紫外-可见吸收光谱仪为Hitachi U-3100,日本;荧光分光光度计为Hitachi F-4600,日本;核磁共振波谱仪为BrukerAvance 400,瑞士;圆二色谱仪为Chirascan,英国。
1.2 化合物NPI1的合成N, N-二甲基胺丙基-1,8-萘二甲酸酐(2a)参照文献合成[27]。1, 8-萘二甲酸酐(1a)(0.6 g,3.0 mmol)和N, N二甲基丙二胺(0.83 g,7.55 mmol)溶于20 mL二氧六环,95 ℃反应20 h,除去溶剂后采用柱色谱纯化,得浅黄色固体2a (0.79 g,2.8 mmol)。
取2a (0.64 g,2.39 mmol)溶于5 mL DMF中,加入4′-(对溴代亚甲基苯基)-2, 2′:6′, 2″-三联吡啶(0.8 g,1.99 mmol),65 ℃避光反应24 h。离心,乙醚洗涤3次,乙醇带干,得白色固体产物NPI1 (1.25 g,1.82 mmol),产率91.8%。
1HNMR(CDCl3,400 MHz):8.68(d,2H,J = 4.4 Hz),8.58(d,2H,J = 8.0 Hz),8.47(s,2H),8.44(d,2H,J = 7.2 Hz),8.02(d,2H,J = 8.0 Hz),7.86(m,4H),7.70(d,2H,J = 8.4 Hz),7.59(m,2H),7.32(m,2H),5.23(s,2H),4.28(m,2H),3.69(m,2H),3.50(s,6H),2.41(m,2H)。MS(MALDI-TOF)[M-Br-]+,604.3,calcd for C39H34N5O2,604.7。
1.3 化合物NPI2的合成N, N-二甲基胺丙基-3-硝基-1,8-萘二甲酸酐(2b)参照文献合成[27]。取3-硝基-1,8-萘二甲酸酐(1b)(0.3 g,1.23 mmol),加入N, N二甲基乙二胺(0.16 g,1.43 mmol),溶于20 mL二氧六环,95 ℃反应20 h,除去溶剂后柱色谱分离,得黄色固体2b (0.32 g,1.03 mmol)。
取2b (0.28 g,0.89 mmol)溶于5 mL DMF中,加入4′-(对溴代亚甲基苯基)-2,2′:6′, 2″-三联吡啶(0.3 g,0.75 mmol),65 ℃下反应24 h。离心,乙醚洗涤3次,乙醇带干,得淡黄色固体产物NPI2 (0.46 g,0.70 mmol),产率93.3%。
1HNMR(DMSO-d6,600 MHz):9.49(d,1H,J = 2.0 Hz),8.94(d,1H,J = 2.0 Hz),8.81(d,1H,J = 8.4 Hz),8.75(d,2H,J = 4.0 Hz),8.69(m,3H),8.64(d,2H,J = 8.0 Hz),8.05(m,5H),7.88(d,2H,J = 8.0 Hz),7.53(m,2H),4.87(s,2H),4.62(m,2H),3.68(m,2H),2.89(s,3H),2.73(s,3H)。MS(MALDI-TOF)[M-Br-]+,635.2,calcd for C38H31N6O4,635.6。
1.4 紫外-可见吸收光谱滴定实验配制30 μmol/L NPI1和NPI2的缓冲溶液(10 mmol/L HEPES,pH = 7.4,0.1 mol/L K+)1.0 mL于1 cm的石英比色皿中,滴加DNA溶液,室温静置10 min。测定萘酰亚胺衍生物NPI1和NPI2与DNA混合溶液的吸收光谱。这些化合物与DNA的结合常数可以通过公式(1) 求得[28]:
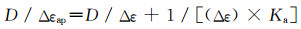
|
(1) |
式中, D为DNA的浓度。Δεap =∣εa-εf︱,Δε =∣εb-εf︱,εa为化合物的表观摩尔消光系数,εa = Aobs/[化合物],即用加入不同浓度DNA后化合物的吸光度值Aobs除以化合物浓度得到。εf为未加入DNA时化合物的摩尔消光系数。εb为所有的化合物与DNA结合后的摩尔消光系数。Ka为化合物与DNA的结合常数。用D/Δεap与D作图,线性拟合后得到的斜率与相应截距的比值为结合常数Ka[29]。
1.5 圆二色谱(CD)实验购买得到的G-四链体溶于缓冲溶液后,以未折叠结构和G-四链体两种形式存在,测定前先将Htelo G-四链体DNA溶液在95 ℃的高温下水浴加热5 min,可促使所有的G-四链体转变为未折叠的Htelo单链结构DNA。然后缓慢冷却至室温,使Htelo单链DNA转变为热力学稳定状态的G-四链体。配制浓度为20 μmol/L的G-四链体DNA缓冲溶液并向其中分别滴加40 μmol/L NPI1和NPI2溶液,4 ℃低温静置过夜,使萘酰亚胺衍生物与G-四链体充分作用。加入比色皿中,在230~320 nm波长范围内扫描CD谱,同时用缓冲溶液作参比液,进行扫描测试。
1.6 荧光共振能量转移(FRET)熔温测定实验将Htelo G-四链体的5′和3′分别采用羧基荧光素(FAM)和6-羧基四甲基罗丹明(TAMRA)标记,得到FHtelo G-四链体(5′-FAM-GGGTTAGGGTTAGGGTTAGGGTAMRA-3′)。在1 mL缓冲溶液(10 mmol/L HEPES,100 mmol/L KCl,90 mmol/L LiCl)中加入NPI1和NPI2 (1.0 μmol/L)与G-四链体FHtelo(0.2 μmol/L)后混合均匀,室温静置2 h。以494 nm为激发波长,测定G-四链体Fhtelo中羧基荧光素和6-羧基四甲基罗丹明荧光基团在505~625 nm范围内的荧光光谱。溶液温度从24 ℃增加至96 ℃,间隔为1 ℃/min,每次恒温保持3 min。将G-四链体FHtelo在523 nm处的荧光强度变化值进行归一化后对温度作图,可求得G-四链体FHtelo的熔解温度[30]。
1.7 分子对接模拟为了进一步了解配合物与G-四链体的结合模式,使用AutoDock 4.2对化合物NPI1和NPI2与G-四链体的作用进行了分子对接模拟[31]。首先通过能量密度泛函(DFT)对化合物NPI1和NPI2的初始结构进行了能量和结构的优化,获得配合物自身的最低能量构象[32]。人体端粒G-四链体的晶体结构模型在蛋白质数据库获得,其PDB文件为1KF1。对AutoDock过程进行了简化处理,将DNA整体结构始终设定为刚性,而允许对接金属配合物有可以转动的键。使用拉马克遗传算法(LGA)对可能的构象和结合位点进行搜索,计算出最有可能的100种构象,从而分析得到配合物与DNA的结合模型和结合能力。
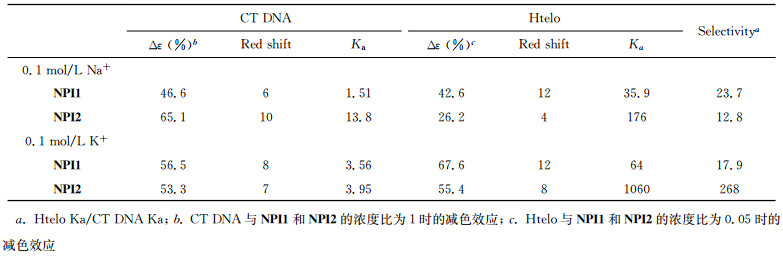
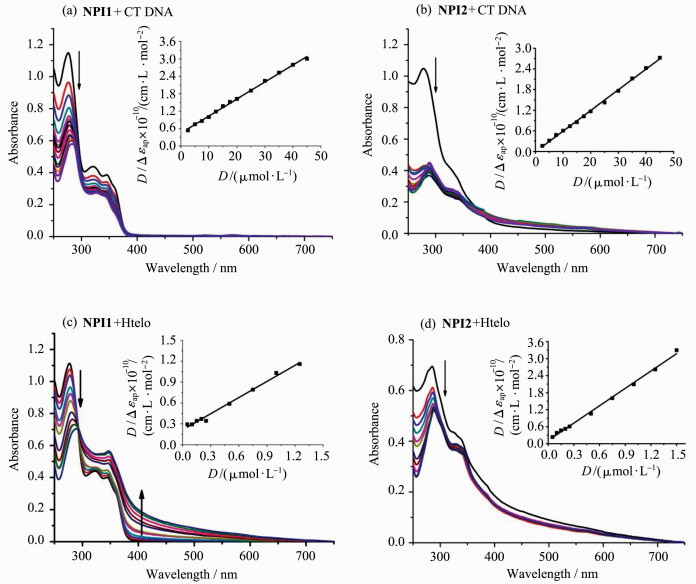
2 结果与讨论 2.1 紫外-可见吸收光谱滴定实验运用紫外-可见吸收光谱滴定实验分别测定不同碱金属离子存在下化合物NPI1和NPI2与双链CT DNA和Htelo G-四链体之间的相互作用模式以及结合能力,并比较了它们对G-四链体选择识别能力的强弱。在含0.1 mol/L Na+的HEPES缓冲溶液中,化合物NPI1和NPI2在278和330 nm处有吸收峰,可归属于三联吡啶和萘酰亚胺的π→π*跃迁吸收(图 2a和b)[33]。加入CT DNA后,NPI1和NPI2的吸收峰强度下降。当CT DNA与NPI1和NPI2的浓度比为1时,278 nm处的减色效应分别为46.6 %和65.1%,吸收峰红移6~10 nm,说明NPI1和NPI2主要以插入方式与CT DNA相结合[34]。利用NPI1和NPI2在277 nm处吸光度的变化与CT DNA浓度作图(图 2a和b),并进行线性拟合,求得NPI1和NPI2与CT DNA的结合常数分别为1.51×105和1.38×106 L/mol(表 1)。
|
图 2 加入CT DNA(a,b)和Htelo G-四链体(c,d)对NPI1和NPI2在含有NaCl(0.1 mol/L)的HEPES缓冲溶液(10 mmol/L,pH 7.4) 中的紫外-可见吸收光谱的影响 箭头表示变化趋势;插图为D/ Δεap对D作图 Fig.2 UV-Vis titration of NPI1 and NPI2 with CT DNA (a, b) and Htelo G-quadruplex (c, d) in HEPES (10 mmol/L, pH 7.4) containing 0.1 mol/L NaCl. The arrow indicates the changes upon addition of DNA Inset: plot of D/ Δεap versus D [NPI1] = 30 μmol/L; [CT DNA] = 0, 2.5, 5, 7.5, 10, 12.5, 15, 17.5, 20, 25, 30, 35, 40, 45 μmol/L |
| 表 1 HEPES缓冲溶液中NPI1、NPI2与CT DNA和Htelo G-四链体的结合常数(Ka,105 L/mol) Table 1 Binding constants of NPI1, NPI2 to CT DNA and Htelo G-quadruplex in HEPES buffer solution(Ka, 105 L/mol) |
在相同条件下加入Htelo G-四链体DNA后,NPI1和NPI2在278 nm处的吸收峰强度下降,当Htelo与NPI1和NPI2浓度比为0.05时,吸收峰红移7~8 nm,减色效应分别为42.6%和26.2%(图 2c和2d)。较大的吸收峰红移和减色效应,表明NPI1和NPI2能够与G-四链体发生末端堆积作用[35]。利用线性拟合作图,求得NPI1与Htelo G-四链体的结合常数为3.59×106 L/mol。化合物NPI1与G-四链体的结合常数是其与CT DNA的23.7倍,说明NPI1对G-四链体作用具有良好的选择性。化合物与双链DNA主要通过DNA碱基平面和沟槽结合,其作用位点相对较少。但是化合物与G-四链体作用时,可与G-四分体、磷酸骨架、沟槽和loop区等多个作用位点结合,其中沟槽区就多达4个[21-23]。NPI1和NPI2中的三联吡啶和萘二酰亚胺基团通过柔性链连接,能够与G-四链体进行多位点结合,这使得NPI1和NPI2与G-四链体的作用要强于双链DNA。考虑到K+是细胞维持正常生理功能必需的离子(在生理状态下,多数细胞的细胞内部的K+离子浓度较高(>100 mmol/L)[36]),所以利用紫外-可见吸收光谱研究了K+存在条件下,化合物NPI1和NPI2与CT DNA和Htelo G-四链体的相互作用,其结合常数见表 1。从表 1可以看出,化合物NPI2与DNA的结合常数要高于NPI1的,这可能是因为具有吸电子作用的硝基与萘酰亚胺在一个平面上,使得硝基取代的萘酰亚胺部分与DNA的碱基具有更强的相互作用[37]。G-四链体的结构表现出多态性。溶液中阳离子不同,会引起G-四链体的连接回路和立体结构发生变化。在K+离子溶液中Htelo G-四链体主要以平行-反平行混合构象存在,而在Na+溶液中Htelo G-四链体主要以反平行结构存在。NPI1和NPI2在K+中与G-四链体的作用比在Na+中的作用强,表明NPI1和NPI2对G-四链体的作用也具有选择性,具体的机理还有待于进一步深入研究[38]。在K+溶液中,NPI2与Htelo G-四链体的结合常数高达1.06 ×108 L/mol,是对双链DNA选择性的268倍,说明NPI2是一种具有良好应用前景的G-四链体稳定剂。
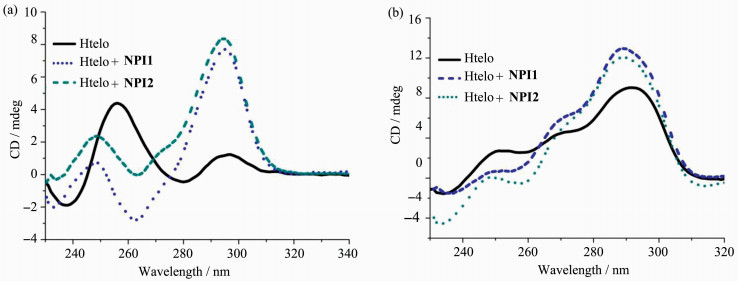
2.2 圆二色谱(CD)实验不同构象的G-四链体具有特征的圆二色谱吸收峰[39, 40],反平行结构G-四链体在295 nm和246 nm处有正峰,而在265 nm处有负峰;平行结构G-四链体在265 nm处有正峰,242 nm处有负峰。采用CD光谱可研究G-四链体与NPI1和NPI2作用前后的结构变化。
在不含碱金属离子的溶液中,Htelo在250 nm和295 nm处显示有两个正峰(图 3a),表明Htelo在该条件下以未折叠结构和G-四链体两种形式存在[41]。加入NPI1和NPI2后,250 nm处的正峰消失,而295 nm处的正峰强度增加,并且在265 nm处出现负峰,表明NPI1和NPI2可诱导Htelo形成反平行结构的G-四链体构象[42]。在K+存在条件下,Htelo在246 nm、265 nm和295 nm处出现正峰,说明Htelo主要以平行-反平行混合结构G-四链体的形式存在[43, 44](图 3b)。加入NPI1和NPI2后峰位未发生明显变化,表明NPI1和NPI2的加入不引起其结构的变化。
|
图 3 (a)不含K+和(b)含有0.1 mol/L K+的Tris-HCl缓冲液中(10 mmol/L,pH 7.4) 加入NPI1和NPI2对Htelo的CD光谱的影响 Fig.3 Effect of NPI1 and NPI2 on the CD spectra of Htelo in Tris-HCl (10 mmol / L, pH 7.4) buffer (a) in the absence of K+ and (b) in the presence of K+ (0.1 mol /L KCl) [NPI1] =[NPI2] =40 μmol/L; [Htelo] = 20 μmol/L |
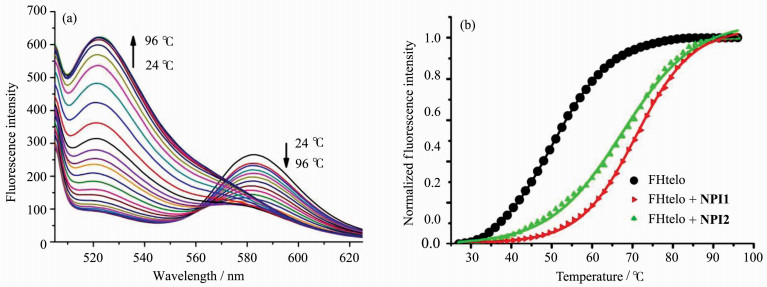
通过FHtelo G-四链体的FRET熔温实验可以进一步研究NPI1和NPI2对端粒G-四链体的稳定作用。FHtelo的5′和3′分别采用羧基荧光素(FAM)和6-羧基四甲基罗丹明进行荧光标记,通过检测羧基荧光素基团的荧光强度变化可以了解加入萘酰亚胺衍生物前后G-四链体熔解温度的变化。与NPI1和NPI2作用后,FHtelo G-四链体的熔解温度分别为70.5 ℃、69.9 ℃,与FHtelo G-四链体的熔解温度(Tm)51.0 ℃相比较,分别升高了19.5 ℃和18.9 ℃(图 4)。表明NPI1和NPI2能够与HteloG-四链体结合并提高其熔解温度,其熔解温度上升幅度与带有正电荷的三联吡啶及其铂配合物相当[24]。
|
图 4 (a)升高温度对NPI2与FHtelo G-四链体混合溶液的荧光光谱的影响;(b) NPI1和NPI2对FHtelo G-四链体熔解温度的影响 Fig.4 (a) Fluorescence spectra of solution of NPI2 and FHtelo upon increasing temperature; (b)effect of NPI1 and NPI2 on the melting temperature FHtelo G-quadruplex [FHtelo] = 0.2 μmol/L; [NPI1] = [NPI2] = 1.0 μmol/L |
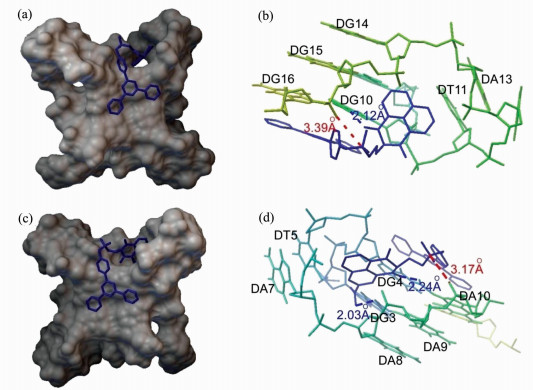
为进一步了解NPI1和NPI2与G-四链体DNA之间的结合模式,采用AutoDock对NPI1和NPI2与G-四链体DNA之间的作用进行分子对接模拟[45, 46]。NPI1和NPI2与人类端粒G-四链体在K+条件下得到的晶体结构(PDB ID:1KF1) 的对接结果如图 5所示。
|
图 5 采用Autodock 4.2对NPI1 (a,b)和NPI2 (c,d)与端粒G-四链体(PDB ID:1KF1) 进行分子对接得到的最优化构型 红色虚线和蓝色虚线分别表示化合物与G-四链体的静电相互作用和氢键作用 Fig.5 The optimized configuration of NPI1 (a, b) and NPI2 (c, d) with the telomeric G-quadruplex (PDB ID: 1KF1) using Autodock 4.2 The red dotted line and the blue dotted line represent the electrostatic interaction and hydrogen bonding of the compound with the G-quadruplex, respectively |
分子对接结果表明NPI1和NPI2中的三联吡啶基团以末端堆积的方式与G-四链体作用,而萘酰亚胺基团以沟槽作用与G-四链体结合(图 5)。NPI1和NPI2中的季铵盐N原子与G-四链体的磷酸骨架的距离为0.3 nm左右,表明化合物与G-四链体间存在静电作用[47]。同时NPI1和NPI2中萘酰亚胺的酰胺C=O与G碱基间存在氢键作用,而NPI2中的硝基与端粒G-四链体的G碱基间也存在氢键作用。所以钾离子存在的溶液中NPI2与Htelo G-四链体具有较高结合常数。NPI1和NPI2可以通过堆积模式、静电作用和氢键作用等多种方式与端粒G-四链体结合,提高了它们与G-四链体的结合能力(Ka>107 L/mol),因此NPI1和NPI2对G-四链体有很好的稳定作用(ΔTm>18 ℃)。
3 结论本文设计合成了两种三联吡啶修饰的萘酰亚胺衍生物NPI1和NPI2并研究了它们与CT DNA及G-四链体DNA的相互作用。研究结果表明NPI1和NPI2对G-四链体有较高的亲和性和选择性,在萘酰亚胺环的3位上引入硝基取代基有利于增强与G-四链体的作用。在不含碱金属离子的条件下,NPI1和NPI2可诱导形成反平行结构的G-四链体;而在K+存在下,G-四链体以混合型G-四链体结构存在。NPI1和NPI2通过堆积模式、静电作用和氢键作用等多种方式与端粒G-四链体结合,因而能够稳定G-四链体,并提高其熔解温度。
| [1] | Hsiang Y H, Liu L F. Identification of mammalian DNA topoisomerase I as an intracellular target of the anticancer drug camptothecin[J]. Cancer Research, 1988, 48(7): 1722–1726. |
| [2] | Hurley L H, Boyd F L. DNA as a target for drug action[J]. Trends in Pharmacological Sciences, 1988, 9(11): 402–407. DOI:10.1016/0165-6147(88)90067-3 |
| [3] | Jaramillo D, Wheate N J, Ralph S F, Howard W A, Tor Y, Aldrich-Wright J R. Polyamide platinum anticancer complexes designed to target specific DNA sequences[J]. Inorganic Chemistry, 2006, 45(15): 6004–6013. DOI:10.1021/ic060383n |
| [4] | Broggini M, Coley H M, Mongelli N, Pesenti E, Wyatt M D, Hartley J A, D'Incalci M. DNA sequence-specific adenine alkylation by the novel antitumor drug tallimustine (FCE 24517), a benzoyl nitrogen mustard derivative of distamycin[J]. Nucleic Acids Research, 1995, 23(1): 81–87. DOI:10.1093/nar/23.1.81 |
| [5] | Janssen S, Durussel T, Laemmli U K. Chromatin opening of DNA satellites by targeted sequence-specific drugs[J]. Molecular Cell, 2000, 6(5): 999–1011. DOI:10.1016/S1097-2765(00)00099-X |
| [6] | Blackbum E H. Switching and signaling at the telomere[J]. Cell, 2001, 106(6): 661–673. DOI:10.1016/S0092-8674(01)00492-5 |
| [7] | Blackburn E H, Greider C W. Identification of a specific telomere terminal transferase activity in tetrahymenaextracts[J]. Cell, 1985, 43(2): 405–413. DOI:10.1016/0092-8674(85)90170-9 |
| [8] | Zakian V A. Telomeres: beginning to understand the end[J]. Science, 1995, 270(5242): 1601–1607. DOI:10.1126/science.270.5242.1601 |
| [9] | Georgiades S, Abd Karim N, Suntharalingam K, Vilar R. Interaction of metal complexes with G-quadruplex DNA[J]. Angewandte Chemie International Edition, 2010, 49(24): 4020–4034. DOI:10.1002/anie.200906363 |
| [10] | Whitehead J P, Lippard S J. Proteins that bind to and mediate the biological activity of platinum anticancer drug-DNA adducts[J]. Metal Ions in Biological Systems, 1996, 32(32): 687–726. |
| [11] | Shay J W, Wright W E. Telomerase activity in human cancer[J]. Current Opinion in Oncology, 1996, 8(1): 66–71. DOI:10.1097/00001622-199601000-00012 |
| [12] | Zahler A M, Williamson J R, Cech T R, Prescott D M. Inhibition of telomerase by G-quartet DNA structures[J]. Nature, 1991, 350(6320): 718–720. DOI:10.1038/350718a0 |
| [13] | McLuckie K I, Di A M, Zecchini H, Xian J, Caldas C, Krippendorff B F, Tannahill D, Lowe C, Balasubramanian S. G-quadruplex DNA as a molecular target for induced synthetic lethality in cancer cells[J]. Journal of the Ame-rican Chemical Society, 2013, 135(26): 9640–9643. DOI:10.1021/ja404868t |
| [14] | Wang Z F, Li M H, Chen W W, Hsu S T, Chang T C. A novel transition pathway of ligand-induced topological conversion from hybrid forms to parallel forms of human telomeric G-quadruplexes[J]. Nucleic Acids Research, 2016, 44(8): 3958–3968. DOI:10.1093/nar/gkw145 |
| [15] | Tawani A, Amanullah A, Mishra A, Kumar A. Evidences for piperine inhibiting cancer by targeting human G-quadruplexdna sequences[J]. Scientific Reports, 2016. DOI:10.1038/srep39239 |
| [16] | Murat P, Balasubramanian S. Existence and consequences of G-quadruplex structures in DNA[J]. Current Opinion in Genetics Development, 2014, 25(4): 22–29. |
| [17] | Mashimo T, Yagi H, Sannohe Y, Rajendran A, Sugiyama H. Folding pathways of human telomeric type-1 and type-2 G-quadruplexstructures[J]. Journal of the American Chemical Society, 2010, 132(42): 14910–14918. DOI:10.1021/ja105806u |
| [18] | Brana M F, Casarrubios L, Dominguez G, Fernández C, Perez J M, Quiroga A G, Navarro-Ranninger C, de Pascual-Teresa B. Synthesis, cytotoxic activities and proposed mode of binding of a series of bis([(9-oxo-9, 10-dihydroacridine-4-carbonyl)amino]alkyl) alkylamines[J]. European Journal of Medicinal Chemistry, 2002, 37(4): 301–313. DOI:10.1016/S0223-5234(02)01348-X |
| [19] | Balasubramanian S, Hurley L H, Neidle S. Targeting G-quadruplexes in gene promoters: a novel anticancer strategy[J]. Nature Reviews Drug Discovery, 2011, 10(4): 261–275. DOI:10.1038/nrd3428 |
| [20] | Drygin D, Siddiqui-Jain A, O'Brien S, Schwaebe M, Lin A, Bliesath J, Ho C B, Proffitt C, Trent K, Whitten J P, Lim J K, Von Hoff D, Anderes K, Rice W G. Anticancer activity of CX-3543: a direct inhibitor of rRNA biogenesis[J]. Cancer Research, 2009, 69(19): 7653–7661. DOI:10.1158/0008-5472.CAN-09-1304 |
| [21] |
张秀凤, 向俊锋, 田明月, 唐亚林. 基于DNA G-四链体识别的抗肿瘤分子筛选及结构设计研究进展[J]. 科学通报, 2009, 54(10): 1374–1386.
Zhang X F, Xiang J F, Tian M Y, Tang Y L. Progress on G-quadruplex as targets in anticancer drug structure screening and rational design[J]. Chinese Science Bulletin, 2009, 54(10): 1374–1386. |
| [22] |
郑小辉, 穆舸, 谭彩萍, 曹乾, 毛宗万. G-四链体DNA稳定剂的研究进展[J]. 中国科学:化学, 2014, 44(4): 484–494.
Zheng X H, Mu G, Tan C P, Cao Q, Mao Z W. The study of G-quadruplexstabilizers[J]. ScientiaSinica: Chemistry, 2014, 44(4): 484–494. |
| [23] | De Rache A, Gueddouda N M, Bourdoncle A, Hommes P, Reissig H, Mergny J. A flexible terpyridine derivative interacts specifically with G-quadruplexes[J]. Chemistry A European Journal, 2016, 22(36): 12651–12654. DOI:10.1002/chem.v22.36 |
| [24] | Wang J T, Li Y, Tan J H, Ji L N, Mao Z W. Platinum(Ⅱ)-triarylpyridines complexes with electropositive pendants as efficient G-quadruplexbinders[J]. Dalton Transactions, 2011, 40(3): 564–566. DOI:10.1039/C0DT01161D |
| [25] | Ou Z Z, Qian Y M, Gao Y Y, Wang Y Q, Yang G Q, Li Y, Jiang K Y. Photophysical, G-quadruplex DNA binding and cytotoxic properties of terpyridine complexes with a naphthalimide ligand[J]. Rsc Advances, 2016, 6(43): 36923–36931. DOI:10.1039/C6RA01441K |
| [26] | Pan D, Wu L Z, Tung C H, Zhang L P. Synthesis of 1, 3-terpyridyl-substituted calixarene ligand and ion recognition[J]. Acta Chimica Sinica, 64(14): 1479–1482. |
| [27] | Stevenson K A, Yen S F, Yang N C, Boykin D W, Wilson W D. A substituent constant analysis of the interaction of substituted naphthalene monoimides with DNA[J]. Journal of Medicinal Chemistry, 1984, 27(12): 1677–1682. DOI:10.1021/jm00378a026 |
| [28] | Wolfe A, Jr S G, Meehan T. Polycyclic aromatic hydrocarbons physically intercalate into duplex regions of denatured DNA[J]. Biochemistry, 1987, 26(20): 6392–6396. DOI:10.1021/bi00394a013 |
| [29] |
欧植泽, 陈晨, 高云燕, 曹璐, 刘桂霞, 李慧珍. 色胺修饰竹红菌素及其稀土离子配位聚合物与DNA相互作用研究[J]. 影像科学与光化学, 2013, 31(5): 361–374.
Ou Z Z, Chen C, Gao Y Y, Cao L, Liu G X, Li H Z. Studies on the interaction of DNA with tryptamine modified hypocrellin and its rare earth metal coordination polymers[J]. Imaging Science and Photochemistry, 2013, 31(5): 361–374. DOI:10.7517/j.issn.1674-0475.2013.05.361 |
| [30] | Yang P, De C A, Teuladefichou M P, Mergny J L, Monchaud D. Engineering bisquinolinium/thiazole orange conjugates for fluorescent sensing of G-quadruplex DNA[J]. Angewandte Chemie International Edition, 2009, 48(12): 2188–2191. DOI:10.1002/anie.v48:12 |
| [31] | Trott O, Olson A J. AutoDockVina: improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading[J]. Journal of Computational Chemistry, 2010, 31(2): 455–461. |
| [32] | Cogoi S, Shchekotikhin A E, Membrino A, Sinkevich Y B, Xodo L E. Guanidino anthrathiophenediones as G-quadruplex binders: uptake, intracellular localization, and anti-Harvey-Ras gene activity in bladder cancer cells[J]. Journal of Medicinal Chemistry, 2013, 56(7): 2764–2778. DOI:10.1021/jm3019063 |
| [33] | Suntharalingam K, White A J, Vilar R. Two metals are better than one: investigations on the interactions between dinuclear metal complexes and quadruplex DNA[J]. Inorganic Chemistry, 2010, 49(18): 8371–8380. DOI:10.1021/ic100884p |
| [34] | Banerjee S, Veale E B, Phelan C M, Murphy S A, Tocci G M, Gillespie L J, Frimannsson D O, Kelly J M, Gunnlaugsson T. Recent advances in the development of 1, 8-naphthalimide based DNA targeting binders, anticancer and fluorescent cellular imaging agents[J]. Chemical Society Reviews, 2013, 42(4): 1601–1618. DOI:10.1039/c2cs35467e |
| [35] | Bertrand H, Monchaud D, Cian A D, Guillot R, Mergny J L, Teuladefichou M P. The importance of metal geometry in the recognition of g-quadruplex-dna by metal-terpyridine complexes[J]. Organic Biomolecular Chemistry, 2007, 5(16): 2555–2559. DOI:10.1039/b708635k |
| [36] | Ueda S, Oiki S, Okada Y. Oscillations of cytoplasmic concentrations of Ca2+ and K+ in fused L cells[J]. The Journal of Membrane Biology, 1986, 91(1): 65–72. DOI:10.1007/BF01870215 |
| [37] | Stevenson K A, Yen S F, Yang N C, Boykin D W, Wilson W D. A substituent constant analysis of the interaction of substituted naphthalene monoimides with DNA[J]. Chemischer Informationsdienst, 1984, 27(12): 1677–1682. |
| [38] | Smargiasso N, Rosu F, Hsia W, Colson P, Baker E S, Bowers M T. G-quadruplex DNA assemblies: loop length, cation identity, and multimer formation[J]. Journal of the American Chemical Society, 2008, 130(31): 10208–10216. DOI:10.1021/ja801535e |
| [39] | Shi S, Liu J, Yao T, Geng X, Jiang L, Yang Q, Cheng L, Ji L. Promoting the formation and stabilization of G-quadruplex by dinuclearRuⅡ complex Ru2(obip)L4[J]. Inorganic Chemistry, 2008, 47(8): 2910–2912. DOI:10.1021/ic7021209 |
| [40] |
孙红霞, 向俊锋, 张亚周, 徐广智, 徐樑华, 唐亚林. 光谱法研究G-四链体与亚甲蓝的相互作用[J]. 科学通报, 2006, 51(9): 1022–1026.
Song H X, Xiang J F, Zhang Y Z, Xu G Z, Xu L H, Tang Y L. Spectroscopic studies of the interaction between methylene blue and G-quadruplex[J]. Chinese Science Bulletin, 2006, 51(9): 1022–1026. |
| [41] | Kypr J, Vorlickova M. Circular dichroism spectroscopy reveals invariant conformation of guanine runs in DNA[J]. Biopolymers, 2002, 67(4-5): 275–277. DOI:10.1002/(ISSN)1097-0282 |
| [42] | Ou Z, Feng Z, Liu G, Chen Y, Gao Y, Li Y, Wang X. Synthesis and g-quadruplex-binding properties of cationic platinum(Ⅱ) terpyridine complexes containing σ-alkynyl auxiliaries[J]. Chemistry Letters, 2015, 44(4): 425–427. DOI:10.1246/cl.141045 |
| [43] | Paramasivan S, Rujan I, Bolton P H. Circular dichroism of quadruplex DNAs: applications to structure, cation effects and ligand binding[J]. Methods, 2007, 43(4): 324–331. DOI:10.1016/j.ymeth.2007.02.009 |
| [44] | Giraldo R, Rhodes D. Promotion of parallel dnaquadruplexes by a yeast telomere binding protein: a circular dichroismstudy[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(16): 7658–7662. DOI:10.1073/pnas.91.16.7658 |
| [45] | Cui X, Lin S, Yuan G. Spectroscopic probing of recognition of the g-quadruplex in c-kit promoter by small-molecule natural products[J]. International Journal of Biological Macromolecules, 2012, 50(4): 996–1001. DOI:10.1016/j.ijbiomac.2012.02.029 |
| [46] | Huey R, Morris G M, Olson A J, Goodsell D S. A semiempirical free energy force field with charge-based desolvation[J]. Journal of Computational Chemistry, 2007, 28(6): 1145–1152. DOI:10.1002/(ISSN)1096-987X |
| [47] | Ghosh S, Mendoza O, Cubo L, Rosu F, Gabelica V, White A J P, Vilar R. Assembly of Palladium(Ⅱ) and Platinum(Ⅱ) metallo-rectangles with a guanosine-substituted terpyridine and study of their interactions with quadruplex DNA[J]. Chemistry A European Journal, 2014, 20(16): 4772–4779. DOI:10.1002/chem.v20.16 |